Recombinación no homóloga
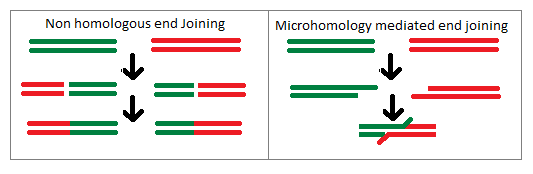
La recombinación no homóloga o recombinación ilegítima, es el proceso mediante el cual se unen dos segmentos bicatenarios de ADN no relacionados. Esta inserción de material genético que no está destinado a ser adyacente tiende a provocar que los genes se rompan, lo que hace que la proteína que codifican no se exprese correctamente. Una de las vías principales por las que esto ocurrirá es el mecanismo de reparación conocido como unión de extremos no homólogos (NHEJ).[1][2]
Descubrimiento
[editar]La recombinación ilegítima es un proceso natural que se descubrió por primera vez que estaba presente en E. coli. Se encontró que un segmento de ADN de 700-1400 pares de bases se había insertado en los operones gal y lac dando como resultado una fuerte mutación polar.[3] Luego se descubrió que este mecanismo tiene la capacidad de insertar otras secuencias genéticas cortas en otras ubicaciones dentro del genoma bacteriano, lo que a menudo conduce a un cambio en la expresión de genes vecinos. A menudo lleva a que los genes vecinos simplemente se apaguen. Sin embargo, algunos de estos segmentos también tenían fuertes señales de inicio y detención que cambiaron la regulación de genes vecinos, lo que provocó cambios en la cantidad de transcripción. Lo que diferenciaba esta forma de recombinación genética de las dependientes de la homología genética era que el proceso observado como ilegítimo no requería el uso de segmentos homólogos de ADN. Si bien no se entendió por completo en ese momento, se reconoció que tenía potencial para generar cambios en la evolución cromosómica.[4]
Mecanismo
[editar]En procariotas
[editar]En los procariotas, la recombinación ilegítima da como resultado una mutación de la secuencia genética del procariota. Este proceso toma diferentes formas en eucariotas, una de las cuales son las deleciones. En una mutación por deleción, el organismo procariótico sufre una recombinación ilegítima que da como resultado la eliminación de un segmento continuo de código genético. Sin embargo, esta forma de mutación ocurre con poca frecuencia entre mutantes de origen natural en lugar de aquellos que han sido inducidos. La recombinación ilegítima también puede ocurrir debido a la transducción de material genético de un bacteriófago. En este caso, un bacteriófago insertará su material genético en una bacteria para insertarlo en el genoma bacteriano en un punto independientemente de la homología genética. Otra forma de recombinación ilegítima en procariotas es la de una duplicación de mutaciones de un genoma. En este caso, una parte del genoma parental se inserta varias veces en el genoma. Esta duplicación inserta el material genético en la misma orientación u opuesta a los segmentos parentales originales, ya que no está dirigida por homología.[4]
En eucariotas
[editar]El mecanismo de recombinación ilegítima es el de la unión de extremos no homólogos en la que dos hebras de ADN que no comparten homología se unirán mediante la maquinaria de reparación de genes. Tras el reconocimiento de una rotura de doble hebra, un complejo proteico mantendrá las dos hebras dentro de la proximidad suficiente para permitir la reparación de las hebras. A continuación, se reparan los extremos del ADN de modo que se eliminen los nucleótidos incorrectos o dañados. Una vez que esto sucede, las hebras se pueden ligar juntas de manera que sean una única hebra de ADN que anteriormente no había sido adyacente. Este proceso es común para las células eucariotas y tiende a actuar como un mecanismo de reparación, pero puede conducir a estas mutaciones si ocurre una recombinación ilegítima. La recombinación ilegítima a menudo tomará la forma de grandes aberraciones cromosómicas dentro de un organismo eucariota, ya que tiene segmentos de ADN mucho más grandes que las células procariotas. Como tal, la unión de extremos no homólogos puede causar una recombinación ilegítima que crea mutaciones de inserción y deleción en los cromosomas, así como la translocación de un segmento cromosómico al de otro cromosoma. Estos cambios a gran escala en el cromosoma en organismos eucariotas tienden a tener efectos deletéreos en el organismo en lugar de conferir un tipo de ventaja genética.[5]
Efectos deletéreos sobre los organismos
[editar]La recombinación ilegítima a menudo tiene efectos deletéreos en un organismo, ya que da como resultado un cambio a gran escala en la secuencia genética de un organismo. Estos cambios darán lugar a mutaciones, ya que la unión de ADN que no se basa en la homología colocará los elementos genéticos en lugares en los que no se habían colocado anteriormente. Esto puede alterar la función de genes que pueden ser esenciales para la función de un organismo. En el caso del cáncer, se ha descubierto que los tumores pueden ser el resultado de una recombinación ilegítima que da como resultado la formación de una horquilla que altera la función genética dentro del genoma de las células tumorales.[6]
Aplicaciones
[editar]La recombinación ilegítima es una herramienta que se puede utilizar en el laboratorio y también es una herramienta de investigación útil. La recombinación ilegítima puede generar mutagénesis aleatoria para generar una alteración aleatoria de la secuencia genética de un organismo.[7] La inducción de esta mutagénesis permite el estudio de una secuencia genética mediante la creación de una mutación en un segmento genético alterando la función de ese segmento genético. Esto permite el estudio de la función de los genes mediante el análisis de las diferencias entre mutantes y organismos naturales para interpretar a qué proceso está vinculado un gen.
Referencias
[editar]- ↑ «Nonhomologous end-joining: mechanisms, conservation and relationship to illegitimate recombination.». Molecular Genetics of Recombination. Topics in Current Genetics. Berlin, Heidelberg: Springer. 2006. pp. 487–513. ISBN 978-3-540-71020-2. doi:10.1007/978-3-540-71021-9_17.
- ↑ Wilson, Thomas E. (2007). Aguilera, Andrés, ed. Molecular Genetics of Recombination. Topics in Current Genetics (en inglés). Springer. pp. 487-513. ISBN 978-3-540-71021-9. doi:10.1007/978-3-540-71021-9_17.
- ↑ Sherratt, David (1978-07). «Illegitimate recombination legitimised». Nature (en inglés) 274 (5668): 213-214. ISSN 1476-4687. doi:10.1038/274213a0.
- ↑ a b Weisberg, Robert A.; Adhya, Sankar (1 de diciembre de 1977). «Illegitimate recombination in bacteria and bacteriophage». Annual Review of Genetics 11 (1): 451-473. ISSN 0066-4197. doi:10.1146/annurev.ge.11.120177.002315.
- ↑ Ehrlich, S. D.; Bierne, H.; d'Alençon, E.; Vilette, D.; Petranovic, M.; Noirot, P.; Michel, B. (15 de diciembre de 1993). «Mechanisms of illegitimate recombination». Gene (en inglés) 135 (1): 161-166. ISSN 0378-1119. doi:10.1016/0378-1119(93)90061-7.
- ↑ Zucman-Rossi, J.; Legoix, P.; Victor, J.-M.; Lopez, B.; Thomas, G. (29 de septiembre de 1998). «Chromosome translocation based on illegitimate recombination in human tumors». Proceedings of the National Academy of Sciences (en inglés) 95 (20): 11786-11791. ISSN 0027-8424. PMC 21718. PMID 9751743. doi:10.1073/pnas.95.20.11786.
- ↑ Khattak, Faisal Asghar; Kumar, Ashutosh; Kamal, Elisabeth; Kunisch, Ralph; Lewin, Astrid (11 de septiembre de 2012). «Illegitimate recombination: An efficient method for random mutagenesis in Mycobacterium avium subsp. hominissuis». BMC Microbiology 12 (1): 204. ISSN 1471-2180. PMC 3511198. PMID 22966811. doi:10.1186/1471-2180-12-204.
