Sustitución sinónima
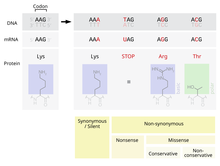
Una sustitución sinónima (a menudo llamada sustitución silenciosa aunque no son siempre silenciosas) es la sustitución evolutiva de una base por otra en el exón de un gen que codifica para una proteína, sin que se produzcan cambios en la secuencia aminoacídica. Esto es posible debido a que el código genético es "degenerado", significando que algunos aminoácidos son codificados por más de un codón; puesto que algunos de los codones para un aminoácido difieren solo por una base de otros que codifican para el mismo aminoácido, una mutación que sustituye a la base "normal" por una de las alternativas resultará en la incorporación del mismo aminoácido en la creciente cadena polipeptídica cuando se traduce el gen. La especificidad del substrato del tRNA al codón distinto puede afectar a la sincronización de la traducción, y a su vez el plegamiento co-translacional de la proteína. [1] Las sustituciones y las mutaciones sinónimas que afectan al ADN no codificante se consideran a menudo mutaciones silenciosas; aun así, no es siempre el caso que la mutación es silenciosa.[1][2][3][4][5] Las mutaciones sinónimas pueden afectar la transcripción, el splicing, el transporte del mRNA, y la traducción, cualquier cambio en estos procesos puede alterar el fenotipo, volviéndose una mutación sinónima no-silenciosa. La especificidad de substrato del tRNA al codón distinto puede afectar a la sincronización de la traducción, y a su vez el plegamiento co-translacional de la proteína. Esto se refleja en el sesgo en el uso de codones que se observa en muchas especies. Una substitución no sinónima da como resultado un cambio en el aminoácido que se puede clasificar arbitrariamente además como conservador (cambio a un aminoácido con las características fisicoquímicas similares), semi-conservador (por ejemplo, un aminoácido negativo por otro positivamente cargado), o radical (aminoácido completamente diferente).
Degeneración del código genético
[editar]La traducción de proteínas envuelve un conjunto de veinte aminoácidos. Cada uno de estos aminoácidos está codificado por una secuencia de tres pares de bases de ADN llamados codón. Debido a que hay 64 codones posibles, pero solo 20 aminoácidos (así como una señal de parada [es decir, los tres codones que no codifican para ningún aminoácido y se conocen como codones de parada], lo que indica que la traducción debe parar), algunos aminoácidos son codificados por 2, 3, 4, o 6 codones diferentes. Por ejemplo, los codones TTT y TTC ambos codifican el aminoácido fenilalanina. Esto se refiere a menudo como redundancia del código genético. Existen dos mecanismos para la redundancia: varios RNAs de transferencia diferentes pueden transportar el mismo aminoácido, o un tRNA puede tener una base no estándar "tambaleante" en la posición tres del anti-codón, la cual reconoce más de una base en el codón.
En el ejemplo antedicho de la fenilalanina, supongamos que la base en la posición 3 del codón TTT fue substituida a C, dejando el codón TTC. El aminoácido en esa posición en la proteína seguirá siendo una fenilalanina. Por lo tanto, es una sustitución sinónima.
Evolución
[editar]Cuando una mutación sinónima o silenciosa ocurre, el cambio se asume a menudo como neutral, significando que no afecta a la aptitud del individuo que lleva el nuevo gen para sobrevivir y para reproducirse.
Los cambios sinónimos pueden no ser neutrales porque ciertos codones se traducen más eficientemente (más rápido y/o más acuradamente) que otros. Por ejemplo, cuando un puñado de cambios sinónimos en el gen alcohol deshidrogenasa en la mosca de la fruta fueron introducidos, cambiando varios codones a los sinónimos subóptimos, la producción de la enzima fue reducida y las moscas de adulto mostraron una tolerancia más baja al etanol.[6][7] En Saccharomyces cerevisiae el uso sinónimo de codones se ha visto que influye en la estabilidad del plegamiento del mRNA, ya que el mRNA codones. [8] Muchos organismos, desde bacterias hasta animales, exhiben el uso sesgado de ciertos codones sinónimos. Este sesgo en el uso de codones puede presentarse por diferentes razones, algunos selectivas y algunos neutras. En Saccharomyces cerevisiae el uso sinónimo de codones se ha visto que influye en la estabilidad del plegamiento del mRNA, ya que el mRNA que codifica distintas estructuras secundarias de la proteína se asocia con la preferencia por determinados codones.[8]
Otra razón por la cual los cambios sinónimos no son siempre neutrales son el hecho de que las secuencias del exón cerca de las fronteras del exón-intrón funcionan como señales de splicing del ARN. Cuando la señal de splicing es destruida por una mutación sinónima, el exón no aparece en la proteína final. Esto da como resultado una proteína truncada. Un estudio encontró que alrededor de un cuarto de variaciones sinónimas que afectan al exón 12 del gen regulador de la conductancia transmembrana de la fibrosis quística, se debe a que ese exón es saltado.[9]
Véase también
[editar]Referencias
[editar]- ↑ «A "Silent" Polymorphism in the MDR1 Gene Changes Substrate Specificity». Science 315 (5811): 525-8. 26 de enero de 2007. PMID 17185560. doi:10.1126/science.1135308.
- ↑ «Hearing silence: non-neutral evolution at synonymous sites in mammals.». Nature Reviews Genetics 7 (2): 98-108. February 2006. PMID 16418745. doi:10.1038/nrg1770.
- ↑ «Synonymous mutations break their silence». Nature Reviews Genetics 8 (2): 92. February 2007. doi:10.1038/nrg2056.
- ↑ «Non-Silent Story on Synonymous Sites in Voltage-Gated Ion Channel Genes». PLoS ONE 7 (10): e48541. 31 de octubre de 2012. PMC 3485311. PMID 23119053. doi:10.1371/journal.pone.0048541.
- ↑ Cooper, David N., ed. (2003). «Single Base Mutation». Nature Encyclopedia of the Human Genome (PDF). MacMillan. ISBN 0333803868.
|editor=y|apellido-editor=redundantes (ayuda) - ↑ Carlini, David B.; Stephan, Wolfgang (2003). «In vivo introduction of unpreferred synonymous codons into the Drosophila Adh gene results in reduced levels of ADH protein». Genetics 163 (1): 239-243. PMC 1462401. PMID 12586711.
- ↑ Carlini, David B. (2004). «Experimental reduction of codon bias in the Drosophila alcohol dehydrogenase gene results in decreased ethanol tolerance of adult flies». Journal of Evolutionary Biology 17 (4): 779-785. PMID 15271077. doi:10.1111/j.1420-9101.2004.00725.x.
- ↑ «Reinvestigating the codon and amino acid usage of S. cerevisiae genome: a new insight from protein secondary structure analysis». Biochem. Biophys. Res. Commun. 354 (3): 693-9. March 2007. PMID 17258174. doi:10.1016/j.bbrc.2007.01.038.
- ↑ Pagani, Franco; Raponi, Michela; Baralle, Francisco E. (2005). «Synonymous mutations in CFTR exon 12 affect splicing and are not neutral in evolution». Proc. Natl. Acad. Sci. U.S.A. 102 (18): 6368-72. PMC 1088389. PMID 15840711. doi:10.1073/pnas.0502288102.
